واکنش زنجیره ای پلیمراز (PCR) و انواع روش PCR
در این مطلب تمام آن چیزی که در رابطه PCR است مانند انواع PCR، مراحل، پارامتر های دخیل و … را برای شما توضیح داده ایم، امیدواریم که برای شما مفید واقع شود. برای کسب اطلاعات بیشتر از مبحث PCR می توانید مطلب بعدی ما که شامل مبحث گزارشگر فلورسنت پروب مورد استفاده در دستگاه های PCR به همراه تجزیه و تحلیل HRM است را، مشاهده کنید. همانطور که مستحضرید شرکت درمان نگار آیندگان شرکت دانش بنیاد و وارد کننده تجهیزات مولکولی الخصوص دستگاه های PCR و Real time PCR می باشد که می توانید از طریق سایت اطلاعات جامع دستگاه ها را مطالعه و برای گرفتن استعلام قیمت با شماره های درج شده در سایت تماس حاصل فرمایید.
PCR چیست؟
واکنش زنجیره ای پلیمراز (Polymerase Chain Reaction) یکی از قدرتمندترین فناوری های زیست شناسی مولکولی می باشد. با این روش، توالیهای خاص درون یک الگوی DNA یا cDNA را میتوان با استفاده از الیگونوکلئوتیدهای خاص (پرایمر)، DNA پلیمراز پایدار در برابر حرارت و چرخه ی حرارتی، هزاران تا میلیون ها بار کپی کرد.
با فرآیند PCR می توان چند مولکول یک اسید نوکلئیک هدف خاص به اندازه یک میکروگرم DNA را تکثیر کرد (درحالیکه این حجم برای تجزیه و تحلیل مستقیم یا استفاده در واکنشهای بیوشیمیایی بسیار کم است).
PCR بطور دقیق، پس از فرآیند تکثیر طبیعی DNA الگوبرداری می شود (سایکی و همکاران، ۱۹۸۵). در فرآیند PCR دو پرایمر الیگونوکلئوتیدی کنار هم قرار گرفته و توالی هدفی را که باید تکثیر شود مشخص می کنند. این پرایمرها با رشته های مخالف با توالی خود در رشته DNA مقابل، هیبرید می شوند تا به عنوان نقطه شروع برای سنتز رشته های DNA جدید عمل کنند. یک DNA پلیمراز مقاوم در برابر حرارت، مانند Taq DNA Polymerase، این سنتز را کاتالیز می کند.
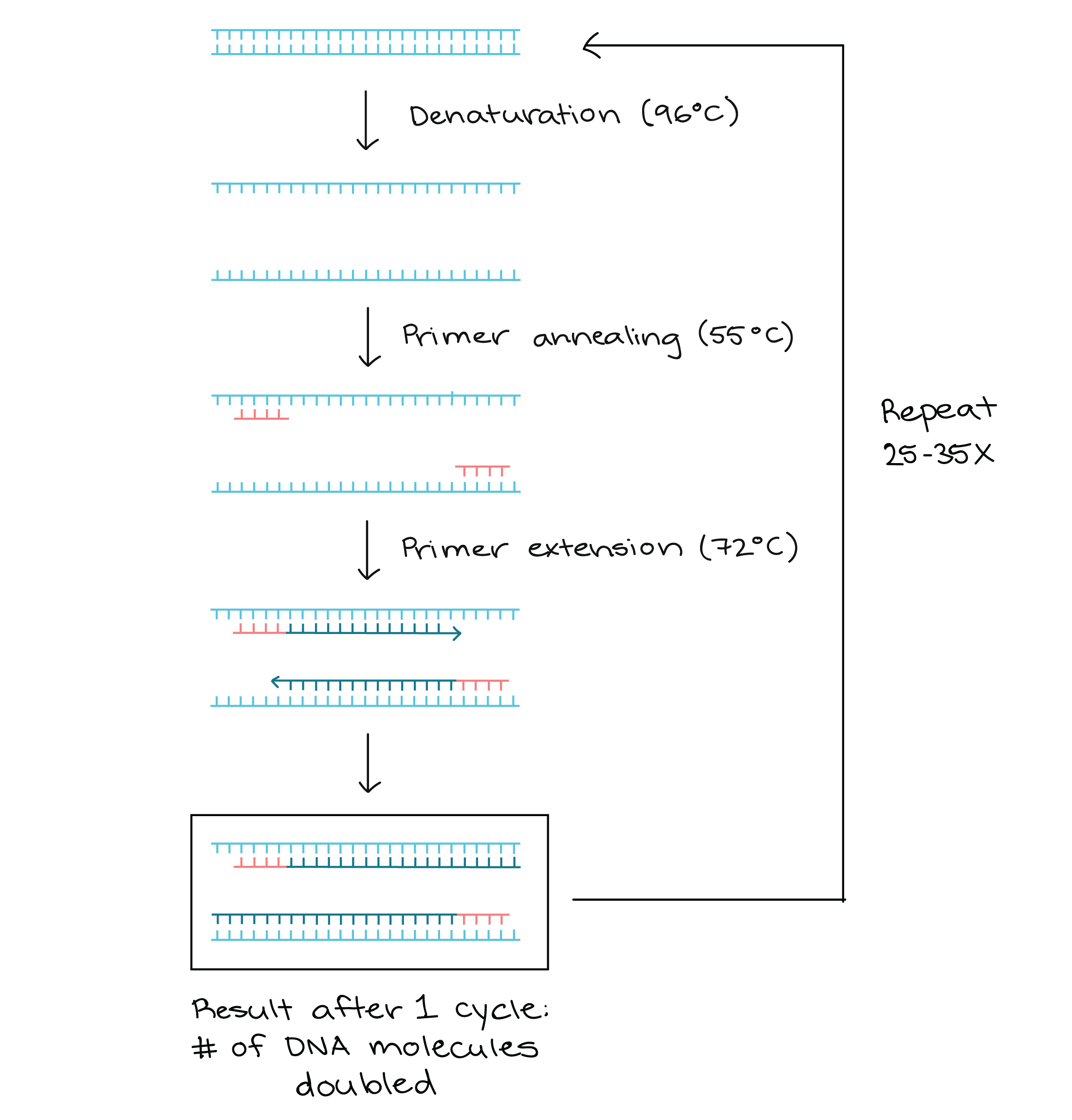
هر چرخه از سنتز PCR (مراحل PCR) شامل سه مرحله است:
- دناتوراسیون (Denaturation)
- بازسرشتگی (Annealing)
- گسترش (Extension)
مرحله ۱ (Denaturation) :
گرما (معمولاً ۹۰ درجه سانتیگراد) DNA دو رشته ای را به دو رشته منفرد جدا می کند. از آنجایی که پیوندهای هیدروژنی پیوند دهنده بازها به یکدیگر ضعیف هستند، در دماهای بالا شکسته می شوند، در حالی که پیوندهای بین دئوکسی ریبوز و فسفات ها، که پیوندهای کووالانسی قوی تری هستند، دست نخورده باقی می مانند. این فرآیند در یک چرخه حرارتی انجام می شود و بطور خودکار چرخه های متناوب گرمایش و سرمایش مورد نیاز برای PCR را کنترل می کند.
مرحله ۲ (Annealing) :
در PCR هدف تکثیر کل رشته DNA نیست، بلکه تکثیر یک توالی هدف تقریباً ۱۰۰-۳۵۰۰۰ جفت باز است که در هر ارگانیسم متفاوت می باشد. پرایمرها انتهای آن توالی هدف را مشخص می کنند.
* (پرایمرها توالی های کوتاه و مصنوعی از DNA تک رشته ای هستند که معمولاً از ۲۰ تا ۳۰ باز تشکیل می شوند.)
انیلینگ معمولاً بین ۴۰ تا ۶۵ درجه سانتیگراد، بسته به طول و توالی پرایمرها انجام می شود و به پرایمرها اجازه می دهد تا بطور خاص به دنباله ی قطعه هدف جهت تکثیر متصل شوند و تکثیر از قطعه هدف انجام شود.
مرحله ۳ (Extension) :
هنگامی که پرایمرها به توالی های DNA مکمل متصل شدند، دما به حدود ۷۲ درجه سانتیگراد افزایش می یابد و یک DNA پلیمراز پایدار در برابر حرارت (به عنوان مثال Taq DNA Polymerase) شروع به سنتز مولکول های DNA دو رشته ای جدید می کند که مشابه DNA هدف اصلی هستند. این کار با کمک dNTPsها که در مسترمیکس آزاد هستند با تسهیل اتصال و اتصال نوکلئوتیدهای مکمل انجام میشود. سنتز همیشه از انتهای ۳′ پرایمر شروع می شود و منحصراً در جهت ‘۵ تا ‘۳ ادامه می یابد. بنابراین، سنتز جدید به طور موثر پرایمرها را گسترش می دهد و یک مولکول مکمل و دو رشته ای از یک الگوی تک رشته ای ایجاد می کند.
پایان چرخه:
در پایان اولین چرخه PCR اکنون دو رشته DNA جدید مشابه هدف اصلی وجود دارد. البته توجه داشته باشید که DNA پلیمراز انتهای توالی هدف را تشخیص نمی دهد. رشته های تازه تشکیل شده دارای یک شروع هستند که دقیقاً با انتهای ‘۵ پرایمر مشخص می شود، اما انتهای ‘۳ دقیقاً مشخص نیست. هر چرخه بعدی این فرآیند کپی را تکرار و چند برابر می کند.
پایان اولین چرخه:
در پایان اولین چرخه، دو نسخه از توالی هدف سنتز شده اند و طول توالی سنتز شده ی DNA از چنین قالبی دارای یک طول دقیقاً تعریف شده است که در انتهای ‘۵ هر یک از دو آغازگر محدود می شود. این رشته های DNA آمپلیکون نامیده می شوند.
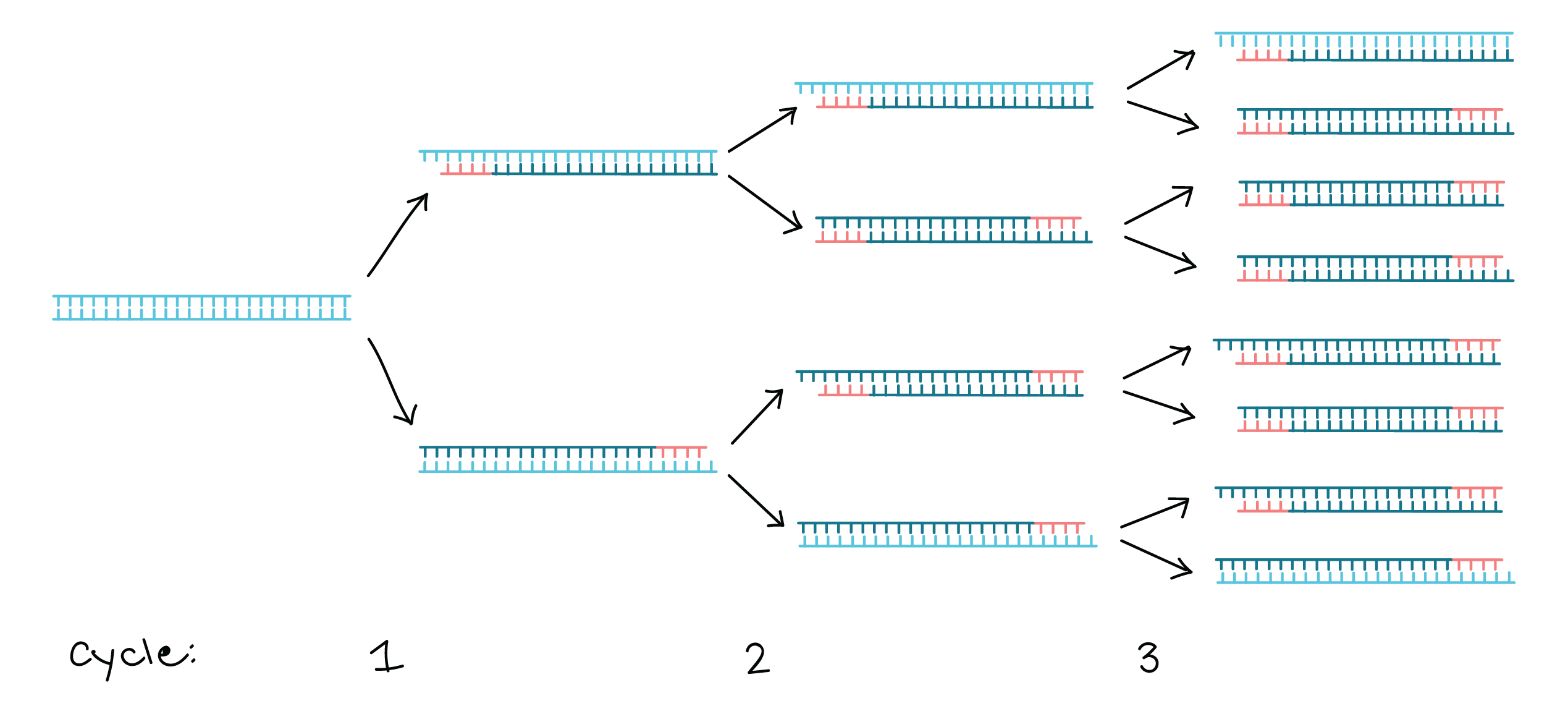
تنها پس از چند چرخه، رشتههای DNA که با توالی هدف مطابقت دارند در تعداد بسیار بیشتری نسبت به توالیهای طول متغیر وجود دارند. به عبارت دیگر توالی کنار یا تعریف شده توسط دو آغازگر، بخشی است که تقویت می شود. این چرخه در فرآیند PCR بارها تکرار می شود (مولیس و فالونا، ۱۹۸۷).
ماهیت تکراری فرآیند PCR، عامل اصلی برتری این روش می باشد. از آنجا که قطعه ی DNA تکثیر شده در یک چرخه بعنوان الگو در چرخه بعدی عمل می کند، تعداد کپی های DNA هدف تقریباً در هر چرخه دو برابر می شود، بنابراین تنها در ۲۰ سیکل، PCR می تواند حدود یک میلیون (۲۲۰) نسخه از هدف را تولید کند.
در PCR سنتی (تشخیص نقطه پایانی) تشخیص و تعیین کمّیت توالی تکثیر شده، در پایان واکنش پس از آخرین چرخه PCR با بررسی محصول حاصله با استفاده از ژل الکتروفورز و همچنین بررسی تصویر باند حاصله انجام می شود. (۱)
Real-Time PCR:
واکنش زنجیرهای پلیمراز در زمان واقعی (Real-Time PCR)، که بعنوان PCR کمّی نیز شناخته میشود، اصلاحی در PCR است که امکان نظارت بر پیشرفت PCR را در زمان واقعی نشان می دهد. علاوه بر بهبود دقت، حسّاسیت و سرعت نیز از مزایای اصلی Real-Time PCR نسبت به PCR پایه است. این تکنیک یک رابطه کمّی قابل اعتماد بین تعداد توالی هدف شروع، قبل از تکثیر با مقدار آمپلیکون انباشته شده در انتهای یک چرخه است.
این روش برای تعیین کمّیت دقیق اسیدهای نوکلئیک هدف، برای تعیین کمّیت mRNA در تجزیه و تحلیل بیان ژن و تعیین بار ویروسی یک نمونه بالینی، بسیار مهم است. علاوه بر این، در این روش نیازی به فرآیندهای پس از PCR مانند الکتروفورز نیست، بنابراین احتمال آلودگی متقاطع ناشی از آمپلیکونهای قبلی به حداقل میرسد. بنابراین این تکنیک انقلابی در تشخیص و تعیین کمّیت اسیدهای نوکلئیک هدف ایجاد کرده و طیف وسیعی از کاربردها را به دست آورده است.
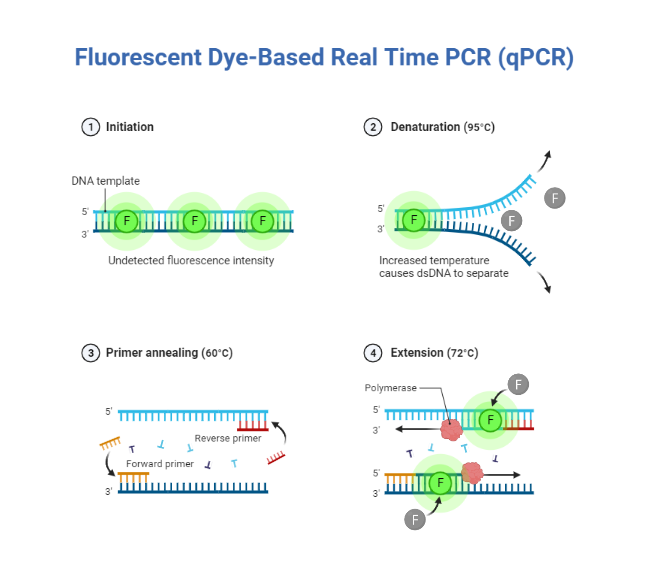
در Real-time PCR، مقدار DNA پس از هر چرخه از طریق رنگهای فلورسنت اندازهگیری میشود که سیگنال فلورسنت افزایشی را در نسبت مستقیم با تعداد مولکولهای محصول PCR (امپلیکون) تولید میکنند و دادههای جمعآوری شده در فاز نمایی واکنش، اطلاعات کمّی در مورد مقدار تکثیر شده ی الگوی هدف بدست میدهد. گزارشگرهای فلورسنت مورد استفاده در Real-time PCR شامل رنگهای متصل شونده به DNA دو رشتهای (dsDNA) یا مولکولهای متصل شونده به پرایمرها یا پروبهای PCR هستند که در طول تکثیر با محصول PCR هیبرید میشوند.
تغییر در فلورسانس در طول واکنش، توسط ابزاری که قابلیت اسکن این تغییرات در چرخه ی دمایی را دارد اندازه گیری می شود. با ترسیم فلورسانس در برابر تعداد چرخه، دستگاه Real-Time PCR یک نمودار تکثیری ایجاد می کند که نشان دهنده تجمع محصول در طول مدت واکنش است.
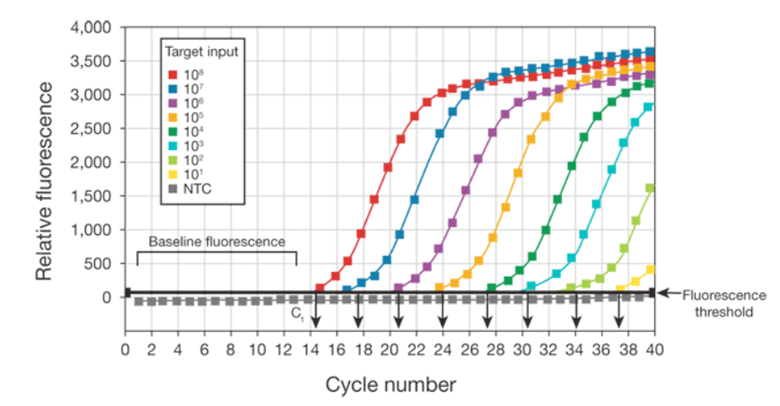
در شکل بالا فلورسانس نسبی در مقابل تعداد چرخه نشان داده شده است. در این نمودار سیگنال فلورسنت از هر نمونه بر اساس شماره سیکل ترسیم شده است. در واقع نمودارهای تکثیر نشان دهنده تجمع محصول در طول مدت آزمایش Real Time PCR می باشد. نمونه های مورد استفاده برای ایجاد نمودارها با رقت های متفاوت از توالی DNA هدف می باشند.
در واقع Real-time PCR نوعی از تکنیک استاندارد PCR است که معمولاً برای تعیین کمّیت DNA یا RNA در یک نمونه استفاده می شود با استفاده از آغازگرهای اختصاصی توالی، می توان تعداد کپی های یک توالی DNA یا RNA خاص را تعیین کرد و با اندازه گیری مقدار محصول تکثیر شده در هر مرحله در طول چرخه، کمّی سازی امکان پذیر است. اگر یک توالی خاص (DNA or RNA) در نمونه فراوان باشد، تکثیر قطعه ی هدف در چرخه های اولیه مشاهده می شود، اما اگر مقدار قطعه هدف کمیاب باشد، تکثیر در چرخه های بالاتر اتفاق می افتد.
کمّیت محصول تکثیر شده با استفاده از پروب های فلورسنت یا رنگ های فلورسنت متصل شونده به DNA و ابزارهای Real-Time PCR که فلورسانس را در حین انجام چرخه ی حرارتی مورد نیاز برای واکنش PCR اندازه گیری می کنند، به دست می آید. مراحل Real-time PCR مانند مراحل PCR و در سه مرحله می باشد که هر چرخه را در واکنش Real-time PCR تشکیل می دهد و واکنش ها معمولاً برای ۴۰ سیکل پیش می روند.
پارامترهای دخیل در تکنیک Real-Time PCR:
-
DNA پلیمراز:
DNA پلیمرازها آنزیم هایی هستند که با ترکیب نوکلئوتیدها، بلوک های سازنده DNA و مولکول های DNA را ایجاد می کنند. این آنزیم ها برای همانندسازی DNA ضروری هستند و معمولاً به صورت جفت برای ایجاد دو رشته DNA یکسان از یک مولکول DNA اصلی عمل می کنند. عملکرد PCR اغلب
به DNA پلیمراز مقاوم در برابر حرارت مرتبط است، بنابراین انتخاب آنزیم درست برای موفقیت این روش بسیار مهم است. یکی از عوامل اصلی موثر بر ویژگی PCR این واقعیت است کهTaq DNA پلیمراز دارای فعالیت باقیمانده در دماهای پایین است. پرایمرها می توانند به طور غیر اختصاصی به DNA متصل شوند و به DNA پلیمراز اجازه میدهند محصول غیر اختصاصی را سنتز کنند. مشکل محصولات غیراختصاصی ناشی از پرایمینگ اشتباه را می توان با استفاده از آنزیم «Hot-Start» به حداقل رساند. استفاده از آنزیم hot start تضمین می کند که هیچ DNA پلیمراز فعالی در طول تنظیم واکنش و مرحله اولیه دناتوراسیون DNA فعال نیست.
-
ترانس کریپتاز معکوس:
رونوشت معکوس (RT)، همچنین به عنوان DNA پلیمراز وابسته به RNA شناخته می شود، این آنزیم یک DNA پلیمراز است که RNA تک رشته ای را به DNA رونویسی می کند. این آنزیم قادر است یک DNA مارپیچ دوگانه را پس از رونویسی معکوس RNA در مرحله اول به DNA تک رشت های سنتز کند. ترانس کریپتاز معکوس (Reverse Transcriptase) برای موفقیت qRT-PCR به اندازه DNA پلیمراز حیاتی است. مهم است که RTی را انتخاب کنید که نه تنها بازده بالایی در سنتز
تمام طول قطعه cDNA را ارائه دهد، بلکه در دماهای بالا نیز فعالیت خوبی داشته باشد. عملکرد دمای بالا نیز برای دناتوره سازی RNAیی که ساختار ثانویه تشکیل داده، بسیار مهم است. در qRT-PCR تک مرحله ای، RT که فعالیت خود را در دماهای بالاتر حفظ میکند، به شما این امکان را میدهد که از پرایمرهای اختصاصی ژن (GSP) با دمای ذوب بالا (Tm) استفاده کنید و ویژگی Specificity محصول را افزایش داده و پسزمینه (Background) واکنش را کاهش میدهد.
-
منیزیم:
منیزیم یک کوفاکتور مورد نیاز برای DNA پلیمرازهای مقاوم در برابر حرارت است. Mg2+ در مخلوط PCR باعث تثبیت dsDNA و افزایش Tm می شود. بنابراین غلظت Mg2+ برای کنترل ویژگی واکنش مهم است. غلظت کم Mg2+ نیاز به جفت شدن بازهای دقیق تری در مرحله انیلینگ دارد.
-
تکنیک تجربی خوب:
اهمیت تکنیک آزمایشگاهی خوب را دست کم نگیرید. بهتر است از تجهیزات و محلولهای اختصاصی برای هر مرحله از واکنشها، از تهیه الگو تا آنالیز پس از PCR استفاده شود. استفاده از نوک سمپلرهای فیلتردار مانع انتشار آئروسل می شود و میکروتیوب های دربدار می تواند به کاهش مشکلات آلودگی متقاطع کمک کند. برای به دست آوردن داده های فشرده از تکرارها (در حالت ایده آل، سه تکرار)، یک مخلوط اولیه تهیه کنید که شامل تمام اجزای واکنش به جز
نمونه باشد. استفاده از مخلوط اولیه تعداد مراحل پیپتینگ را کاهش می دهد و در نتیجه احتمال آلودگی چاهک های متقاطع و سایر خطاهای پیپتی را کاهش می دهد.
-
تمپلیت (Template):
برای هر واکنش Real-Time PCR باید از ۱۰ تا ۱۰۰۰ نسخه از اسید نوکلئیک الگو استفاده شود. این مقدار معادل تقریباً ۱۰۰ پیکوگرم تا ۱ میکروگرم DNA ژنومی یا cDNA سنتز شده از ۱ pg تا ۱۰۰ng از RNA کل است. حجم الگوی بیشتر از این مقدار ممکن است سطوح آلودگی بالاتری را به همراه داشته باشد که میتواند کارایی PCR را تا حد زیادی کاهش دهد. بسته به ویژگی پرایمرهای واکنش، ممکن است استفاده از cDNA بجای DNA ژنومی یک گزینه ی بهتر برای کاهش آلودگی باشد. گزینه ی دیگر این است که الگو را با DNase I تریت کنید.RNA دست نخورده و فوق خالص برای سنتز cDNA از تمام طول الگوی هدف و با کیفیت ضروری است و ممکن است برای تعیین کمّیت دقیق mRNA مهم باشد. RNA باید عاری از هرگونه آلودگی RNase باشد. RNA توتال بطور معمول در qRT-PCR به خوبی کار می کند.
-
پرایمرهای مورد استفاده در واکنش Real-Time PCR:
طول آمپلیکون باید تقریباً ۵۰ تا ۱۵۰ جفت باز باشد، زیرا محصولات طولانی تر به اندازهی کارآمد تکثیر نخواهند شد. بطور کلی، پرایمرها باید ۱۸-۲۴ نوکلئوتید داشته باشند. پرایمرها باید بر اساس دستورالعمل های استاندارد PCR طراحی شوند و باید برای دنباله الگوی هدف خاص طراحی شده و فاقد ساختار ثانویه داخلی باشند. پرایمرها نباید حاوی کشش دنباله های هموپلیمر ( مانند پلی (dG)) یا موتیف های تکرار شونده باشند، زیرا ممکن است به طور نامناسب هیبرید شوند. جفت های پرایمر باید دمای ذوب سازگار (در حدود ۵ درجه سانتیگراد) داشته باشند و تقریباً ۵۰٪ محتوای GC باشند. پرایمرهایی با محتوای GC بالا می توانند هیبریدهای ناقص پایدار را تشکیل دهند. برعکس، محتوای بالای AT، Tm هیبریدهای کاملاً منطبق را کاهش می دهد. در صورت امکان، انتهای ‘۳ پرایمر باید غنی از GC باشد تا واکنش تا انیلینگ انتهایی پیش رود. برای qRT-PCR، باید پرایمرهایی طراحی شود که با اگزونهای دو طرف اینترون یا مرز اگزون/اگزون mRNA همپوشانی داشته باشد.
نرم افزار طراحی پرایمر:
برنامههای نرمافزار طراحی پرایمر، مانند طراح ™OligoPerfect، نرمافزارPrimer xpress® و نرمافزار تحلیل توالی، مانند نرمافزار Vector NTI® میتوانند به طور خودکار یک توالی هدف را ارزیابی کرده و پرایمرهایی را برای آن بر اساس معیارهای ذکر شده طراحی کنند. حداقل، استفاده از نرمافزار طراحی پرایمر تضمین میکند که پرایمرها برای دنباله هدف خاص هستند و ساختار ثانویه داخلی ندارند و از هیبریداسیون تکمیلی در انتهای ۳ در هر آغازگر اجتناب می کند. همانطور که قبلا ذکر
شد، طراحی پرایمر خوب به ویژه هنگام استفاده از رنگهای متصل شونده به DNA برای تشخیص آمپلیکون بسیار مهم است.
Real-Time PCR چگونه کار می کند؟
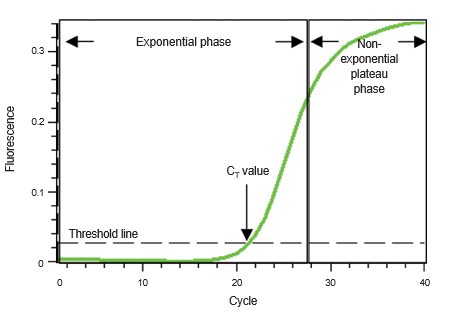
در نمودار زیر، شماره چرخه PCR روی محور x نشان داده شده و فلورسانس حاصل از واکنش تقویت، که متناسب با مقدار محصول تقویت شده در لوله است، در محور y نشان داده شده است. نمودار تقویت دو فاز را نشان میدهد، یک فاز نمایی و به دنبال آن یک فاز فلات غیر نمایی. در طول فاز نمایی، مقدار محصول PCR در هر سیکل تقریباً دو برابر می شود. با ادامه واکنش، اجزای واکنش مصرف میشوند و در نهایت یک یا چند جزء کاهش می یابد.
در این مرحله، واکنش کند می شود و وارد فاز فلات می شود. در ابتدا، فلورسانس در سطوح پسزمینه باقی میماند و افزایش فلورسانس قابل تشخیص نیست (چرخههای ۱ تا ۱۸شکل) حتی اگر محصول به صورت تصاعدی انباشته شود. در نهایت، محصول تکثیر شده کافی برای تولید یک سیگنال فلورسنت قابل تشخیص جمع می شود.
شماره چرخه ای که در آن این اتفاق می افتد، سیکل آستانه یا CT نامیده می شود. از آنجایی که مقدار CT در فاز نمایی زمانی که معرف ها محدود نیستند اندازه گیری می شود، می توان از RT-qPCR برای محاسبه مطمئن و دقیق مقدار اولیه الگوی موجود در واکنش استفاده کرد.
CT یک واکنش عمدتاً با مقدار الگوی موجود در شروع واکنش تکثیر تعیین می شود. اگر مقدار زیادی الگو در شروع واکنش وجود داشته باشد، چرخه های تکثیر نسبتا پایینی برای جمع آوری محصول کافی برای ایجاد یک سیگنال فلورسنت در بالای پس زمینه مورد نیاز خواهد بود. بنابراین، واکنش CT کم یا زودرسی خواهد داشت.
در مقابل، اگر مقدار کمی از الگو در شروع واکنش وجود داشته باشد، چرخه های تقویت بیشتری
برای افزایش سیگنال فلورسنت از پس زمینه مورد نیاز خواهد بود. بنابراین، واکنش یک CT بالا یا دیررس خواهد داشت. این رابطه پایه و اساس جنبه کمّی Real-Time PCR را تشکیل می دهد.
Reverse Transcription PCR:
از نظر تئوری، Real-time PCR تنها می تواند برای تقویت الگوها در قالب مولکول های DNA اعمال شود. پس چگونه می توان یک نمونه RNA را تشخیص داد و کمّیت آن را سنجید؟ برای این منظور، ابتدا مولکول RNA با استفاده از رونوشت معکوس (Reverse Transcriptase: RT) به DNA مکمّل (cDNA) رونویسی می شود و سپس cDNA تک رشت های تولید شده به DNA دو رشت های تبدیل می شود. سپس این DNA دو رشت های با استفاده از PCR استاندارد تکثیر می شود. این روش به عنوان واکنش زنجیرهای پلیمراز رونویسی معکوس در زمان واقعی (RT-PCR) شناخته می شود.
RT-PCR در زمان واقعی می تواند با استفاده از روش یک مرحله ای یا دو مرحله ای انجام شود.
-
RT-PCR یک مرحله ای:
در این فرآیند، مرحله ی RT با PCR جفت می شود و RNA به cDNA معکوس رونویسی شده، سپس در همان واکنش تکثیر می شود. از مزایای اصلی این روش میتوان به سرعت راه اندازی، ارزان بودن و دستکاری کمتر نمونهها برای کاهش خطای پیپت و آلودگی اشاره کرد. با این حال، از آنجایی که این روش از پرایمرهای اختصاصی ژن برای هر دو RT و PCR که در یک لوله واکنش رخ میدهند، استفاده میکند، دیگر ژنهای مورد نظر را نمیتوان برای تجزیه و تحلیل بعدی تکثیر کرد.
-
RT-PCR دو مرحله ای:
این فرآیند از دو مرحله مجزا تشکیل شده است. مرحله اولیه یک واکنش RT برای ساخت cDNA است. مرحله دوم، تقویت cDNA با استفاده از Real-Time PCR سنتی است. مزیت اصلی RT-PCR دو مرحلهای این است که cDNA معمولاً با استفاده از پرایمرهای تصادفی هگزامر یا الیگو-dT تولید میشود که امکان تبدیل کامل پیامهای موجود در نمونه RNA به cDNA را فراهم میکند، بنابراین امکان تجزیه و تحلیل ژنهای دیگر را در آینده فراهم میکند.
پروپ ها:
سیستمهای PCR در زمان واقعی از یک گزارشگر فلورسنت پروب برای تشخیص و تعیین کمّیت استفاده میکنند. بطور کلی پروب ها بسته به نوع فلورسنت مورد استفاده و ویژگی تشخیص PCR به دو گروه اصلی طبقه بندی می شوند:
- رنگ های قابل اتصال به DNA مانند SYBR Green I
- پرایمرها یا پروب های الیگونوکلئوتیدی با توالی خاص نشاندار شده با رنگ (مانند TaqMan وmolecular beacons، پروب های هیبریدیزاسیونی و Eclipes، آمپلی فور، Scorpions، LUX و پروب های BD QZyme
برای مطالعه ادامه مطلب انواع پروب ها و تجزیه و تحلیل HRM، به مطلب بعدی مراجعه کنید.
نویسنده: خانم پاشایی
References:
- I Made Artika, Y. Permata Dewi, I.M. Nainggolan, J.E. Siregar & U. Antonjaya. “Real-Time Polymerase Chain Reaction: Current Techniques, Applications, and Role in COVID-19 Diagnosis”. Genes (Basel). 2022 Dec; 13(12): 2387. Published online 2022 Dec 16. Doi: ۱۰٫۳۳۹۰/genes13122387.
- Real-time PCR handbook. Life Technologies. www.gene-quantification.de
- Real-Time PCR Applications Guide, Bio-Rad:
- Mathews DH et al., Expanded sequence dependence of thermodynamic parameters provides robust prediction of RNA secondary structure, J Mol Biol 288, 910–۹۴۰ (۱۹۹۹))
- Rozen S and Skaletsky HJ, Primer3 on the WWW for general users and for biologist programmers, pp 365–۳۸۶ in Bioinformatics Methods and Protocols: Methods in Molecular Biology (Krawetz S and Misener S, eds), Humana Press, Totowa, NJ (2000); source code available at http://fokker.wi.mit.edu/primer3/
- Zuker M et al., Algorithms and thermodynamics for RNA secondary structure prediction: a practical guide, pp 11–۴۳ in RNA Biochemistry and Biotechnology (Barciszewski J and Clark BFC, eds), Kluwer Academic Publishers, Boston (1999)
- Real-Time PCR Applications Guide. Degen, Hans-Joachim, Deufel, Annette, Eisel, Doris Grünewald-Janho, Stefanie Keesey, Joe. 2006 by Roche Diagnostics GmbH, Mannheim. PCR Applications Manual 3rd edition. www.Roche-Applied-Science.com
- Quantitative Real-Time PCR Methods and Protocols (3rd Edition). Editors: Roberto Biassoni Laboratorio Medicina Molecolare Dipartimento Medicina Traslazionale Istituto Giannina Gaslini Genova, Italy. ISSN 1064-3745 ISSN 1940-6029 (electronic) ISBN 978-1-4939-0732-8 ISBN 978-1-4939-0733-5 (eBook) DOI 10.1007/978-1-4939-0733-5 Springer New York Heidelberg Dordrecht London Library of Congress Control Number: 2014936291 © Springer Science+Business Media New York 2014.