انواع پروب و تجزیه و تحلیل HRM
پروب چیست:
سیستمهای PCR در زمان واقعی (Real time PCR) از یک گزارشگر فلورسنت پروب برای تشخیص و تعیین کمّیت استفاده میکنند. بطور کلی پروب ها بسته به نوع فلورسنت مورد استفاده و ویژگی تشخیص PCR به دو گروه اصلی طبقه بندی می شوند:
- رنگ های قابل اتصال به DNA مانند SYBR Green I
- پرایمرها یا پروب های الیگونوکلئوتیدی با توالی خاص نشاندار شده با رنگ (مانند TaqMan وmolecular beacons، پروب های هیبریدیزاسیونی و Eclipes، آمپلی فور، Scorpions، LUX و پروب های BD QZyme
متداولترین رنگ های مورد استفاده برای PCR در زمان واقعی، پروبهای هیدرولیز رنگی SYBR Green I و TaqMan هستند که امکان تشخیص آمپلیکون های خاص و غیر اختصاصی را می دهند. برای گروه دوم، فلوروفورها به الیگونوکلئوتیدها متصل می شوند، بنابراین آنها فقط آمپلیکون های خاص را شناسایی می کنند.
پروبی که برای سنجش qPCR انتخاب میکنید به کاربرد شما در تست بستگی دارد، اینکه آیا در حال انجام واکنشهای Single plex یا Multi plex هستید و با توجه هزینه ی تست، انتخاب پروب ها را متفاوت می کند. برای آزمایشهای تک پلکس کم توان، رنگهای اتصال به DNA ممکن است ترجیح داده شوند زیرا طراحی این نشانگرها در ابتدا مقرون به صرفهتر، آسانتر و تنظیم سریعتر هستند. با این حال، برای آزمایشهای با توان بالا، یک آزمایش مبتنی بر پرایمر یا پروب فلورسنت (سینگلپلکس یا مالتی پلکس) ممکن است مطلوبتر باشد زیرا هزینه اولیه را میتوان در بسیاری از آزمایشها تقسیم کرد و قابلیت مالتی پلکس میتواند زمان سنجش را کاهش دهد. سنجش مولتی پلکس به استفاده از نشانگر مبتنی بر پرایمر یا پروب فلورسنت نیاز دارد، زیرا فقدان ویژگی رنگهای متصل شونده به DNA آنها را با سنجشهای مالتی پلکس کمّی ناسازگار میکند.
1.سایبرگرین:
متداول ترین رنگ اتصال به DNA برای PCR در زمان واقعی SYBR Green I است که به طور غیر اختصاصی به DNA دو رشته ای (dsDNA) متصل می شود. SYBR Green I هنگامی که در محلول آزاد است فلورسانس کمی از خود نشان می دهد، اما فلورسانس آن با اتصال به dsDNA تا 1000 برابر افزایش می یابد. بنابراین سیگنال فلورسنت آزاد شده از یک واکنش متناسب با مقدار dsDNA موجود در واکنش می باشد و با تکثیر و بیشتر شدن میزان قطعه ی DNAی هدف افزایش خواهد یافت.
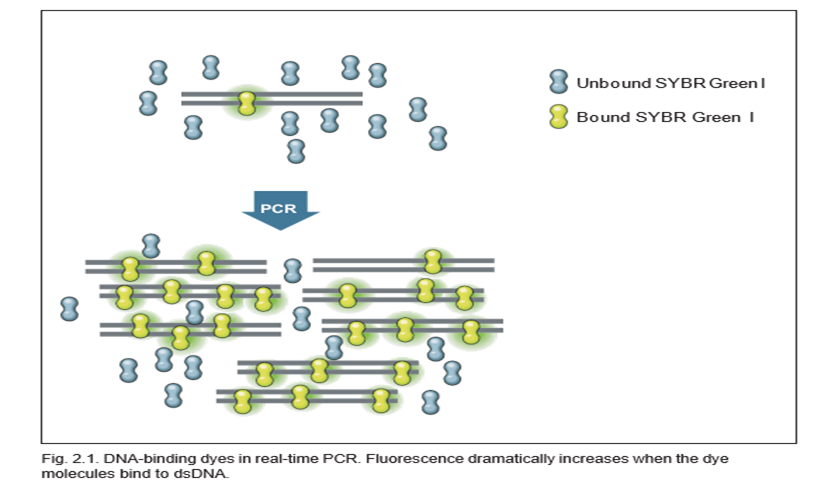
مزایای استفاده از رنگهای متصل شونده به dsDNA:
- طراحی سنجش ساده (فقط دو آغازگر مورد نیاز است؛ طراحی پروب لازم نیست)،
- توانایی آزمایش سریع ژنهای متعدد بدون طراحی پروبهای متعدد
- هزینه اولیه کمتر (هزینه پروب بیشتر)
- توانایی انجام تجزیه و تحلیل منحنی مذاب برای بررسی ویژگی واکنش تکثیری
2.پرایمر و پروب های فلورسنت مبتنی بر شیمی:
پرایمر و پروب های فلورسنت زیادی مبتنی بر مواد شیمیایی ابداع شده اند و از طرف فروشندگان و کمپانی های مختلفی در سراسر دنیا در دسترس هستند. متداولترین آنها، پروب های TaqMan و molecular beacons هستند. این پرایمر و پروب ها یکسری ویژگی های مشترک دارند. بطور کلی، این مواد شیمیایی با استفاده از انتقال انرژی با تشدید فلورسانس (FRET) یا از طریق خاموش کردن فلورسانس موجب می شوند تا فلورسانس خاص فقط در حضور محصول تکثیر شده تشخیص داده شود.
پرایمر یا پروب های الیگونوکلئوتیدی برای یک تارگت یا قطعه ی خاص طوری طراحی می شود که، حاوی یک گزارشگر (ریپورتر) فلوروفور باشد. عملکرد این الیگونوکلئوتید بطوری می باشد که فلورسانس تا زمانی که به الگوی هدف خاص متصل نشود خاموش باقی می ماند و فلورسانسی از خود ساطع نمی کند، که اینکار با اتصال یک مولکول خاموش کننده به کاوشگر و ایجاد مکانیسم خاموش کننده همراه است. هنگامی که کاوشگر به هدف خاص خود متصل می شود مولکول فلورسانس و کوانچر از هم جدا می شوند و گزارشگر شروع به ساطع کردن فلورسانس از خود می کند. مکانیسم چگونگی عملکرد و جداسازی پروب های مختلف در پایین توضیح داده شده است. پرایمرها و پروب های فلورسنت در PCR در زمان واقعی دو مزیت اصلی نسبت به رنگ های متصل شونده به DNA دارند. اول اینکه آنها به طور خاص دنباله هدف را تشخیص می دهند بنابراین محصولات غیر اختصاصی بر دقت کمّیت تأثیر نمی گذارند. دوم اینکه آنها اجازه می دهند واکنش های چندگانه همزمان در یک لوله انجام شود. موارد توضیح داده شده به ترتیب زیر می باشند:
TaqMan Assay
Molecular Beacons
Other Chemistries:
Hybridization probes .a
Eclipse probe .b
Amplifluor chemistry .c
Scorpions’ primer .d
LUX primer assays .e
BD QZyme primers .f
TaqMan Assay .1:
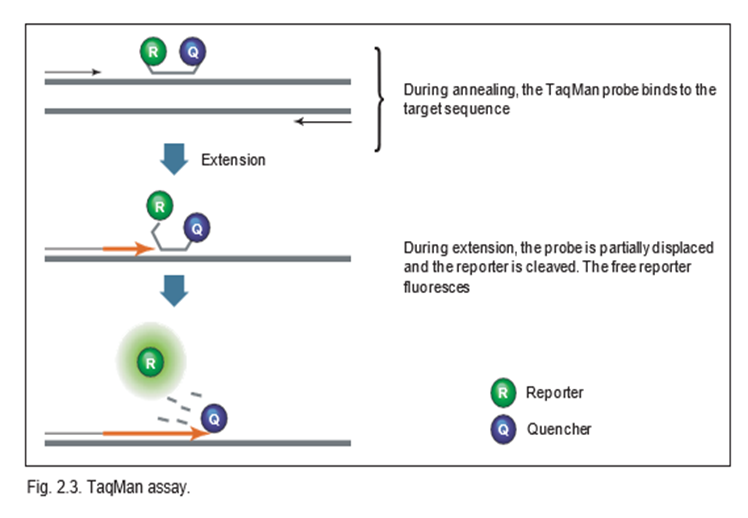
TaqMan Assay توالی خاصی دارد و با یک پروب الیگونوکلئوتیدی فلورسنت نشانه گذاری شده است. شکل زیر نحوهی عملکرد این Assay رانشان می دهد.
همانطور که در شکل مشخص است، این assay بعنوان سنجش ‘5-Nuclease نیز شناخته می شود. سنجش TaqMan از فعالیت exonuclease-‘5 برخی از پلیمرازهای مقاوم در برابر حرارت، مانند Taq یا Tth استفاده می کند.
این کاوشگر حاوی یک گزارشگر فلورسنت در انتهای ‘5 و یک خاموش کننده در انتهای ‘3 است. هنگامیکه دست نخورده است و واکنشی صورت نگرفته، فلورسانس گزارشگر (ریپورتر) به دلیل نزدیکی به کوئنچر خاموش است. در طول مرحله ترکیبی بازپخت/گسترش واکنش تکثیر، پروب با قطعه هدف هیبرید می شود و فعالیت اگزونوکلئاز ‘5 ← ‘3 خاص dsDNA Taq یا Tth با جدا شدن شروع می شود. در نتیجه گزارشگر از کوئنچر جدا می شود و سیگنال فلورسانس متناسب با مقدار محصول تکثیر شده را آزاد می کند. یکی از جفت خاموش کننده های گزارشگر فلورسنت که معمولاً مورد استفاده قرار می گیرد فلورسین (FAM) می باشد که فلورسانس سبز ساطع می کند.
از مزایای اصلی استفاده از پروب های TaqMan میتوان به Specificity بالا، نسبت سیگنال به نویز بالا و توانایی انجام واکنش های چندگانه همزمان اشاره کرد. اما از معایب این assay می توان به هزینه ی اولیه بالای آن اشاره کرد، همچنین طراحی پروب های با کیفیت نیز در چگونگی سنجش بی اهمیت نیست.
Molecular-Beacons .2:
علاوه بر دو آغازگر اختصاصی توالی، سنجشهای Molecular-Beacons از یک کاوشگر الیگونوکلئوتیدی برچسبگذاری شده با فلورسنت خاص دنبالهای استفاده میکند که فانوس مولکولی نامیده میشود، این مولکول یک رنگ با الیگونوکلئوتید لیبل شده (25-40 NT) می باشد، این ساختار بصورت یک حلقه با ساختار ساقه و سنجاق سر می باشد. یک گزارشگر فلورسنت به انتهای 5′ فانوس مولکولی و یک خاموش کننده به انتهای 3′ متصل است. این حلقه به گونه ای طراحی شده است که بطور خاص با یک بخش 15 تا 30 نوکلئوتیدی از توالی هدف هیبرید شود. در دو طرف حلقه 5 تا 6 nt وجود دارد که مکمل یکدیگر هستند و ساختار ساق های را تشکیل می دهند که گزارشگر و خاموش کننده را به هم نزدیک می کند. در ساختار سنجاق سر، به دلیل نزدیکی فیزیکی آن به خاموش کننده، هیچ فلورسانسی از گزارشگر تشخیص داده نمی شود. در طول مرحله بازپخت واکنش تکثیر، فانوس مولکولی به دنباله هدف خود متصل می شود و گزارشگر و خاموش کننده را از هم جدا می کند بطوری که گزارشگر دیگر خاموش نمی شود. برخلاف assayهای TaqMan، این مولکول ها در طول تکثیر جابهجا میشوند اما از بین نمیروند، زیرا از یک DNA پلیمراز فاقد فعالیت اگزونوکلئاز ’5 استفاده میشود. مقدار فلورسانس منتشر شده توسط گزارشگر روی فانوس مولکولی متناسب با مقدار تکثیر شده ی الگوی هدف در واکنش است.
فانوس های مولکولی نسبت به سایر مواد شیمیایی مزایایی دارند که باعث استفاده از آنها برای برخی تست ها می شود، بعنوان مثال آنها بسیار مولکول های خاصی در واکنش هستند و می توانند برای تست های مالتی پلکس استفاده شوند و اگر دنباله هدف دقیقاً با دنباله فانوس مولکولی مطابقت نداشته باشد، هیبریداسیون و فلورسانس رخ نخواهد داد (یک ویژگی مطلوب برای آزمایش های تشخیص آللی).
hd اصلی استفاده از بیکن های مولکولی در طراحی آنها نهفته است. ساقه گیره باید به اندازه کافی قوی باشد که مولکول به طور خود به خود به شکل های غیر سنجاقی که منجر به فلورسانس ناخواسته می شود، جمع نشود. در عین حال، ساقه گیره نباید خیلی قوی باشد، در غیر این صورت ممکن است بیکن های مولکولی بدرستی با هدف هیبرید نشود.
![]()
Other Chemistries .3:
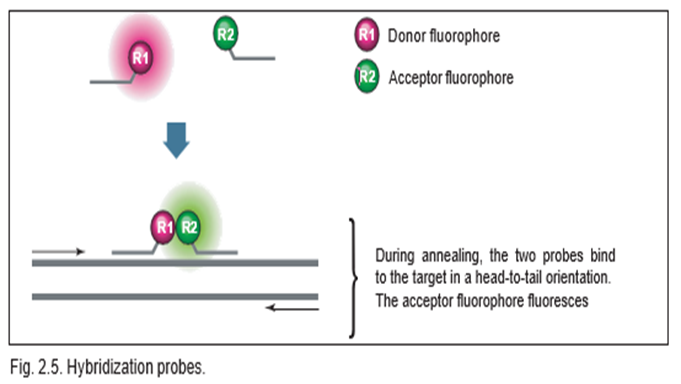
Hybridization probes .a:
نشانگر PCR با پروب هیبریداسیون علاوه بر دو پروب الیگونوکلئوتیدی با توالی اختصاصی، از دو آغازگر با توالی اختصاصی استفاده میکند. همانطور که در شکل زیر نشان داده شده است، این دو نشانگر برای اتصال به توالی های مجاور در الگوی هدف طراحی شدهاند. پروب اول یک رنگ دهنده را در انتهای ‘3 خود حمل می کند، در حالی که پروب دوم یک رنگ پذیرنده را در انتهای ‘5 خود حمل می کند. رنگ دهنده و گیرنده به گونهای انتخاب می شوند که طیف انتشار رنگ دهنده به طور قابل توجهی با طیف تحریک رنگ پذیرنده همپوشانی داشته باشد، از طرفی طیف انتشار رنگ دهنده از طیف انتشار رنگ پذیرنده قابل تفکیک باشد. تحریک در طول موج مخصوص رنگ دهنده انجام می شود و واکنش در طول موج انتشار رنگ پذیرنده کنترل می شود. در طول مرحله PCR (annealing)، پروب ها به ترتیب از سر به دم با دنباله های هدف خود هیبرید می شوند. این امر مولکول های فلورسنت را به هم نزدیک می کند و امکان انتقال انرژی تشدید فلورسانس از دهنده به گیرنده را فراهم می کند.
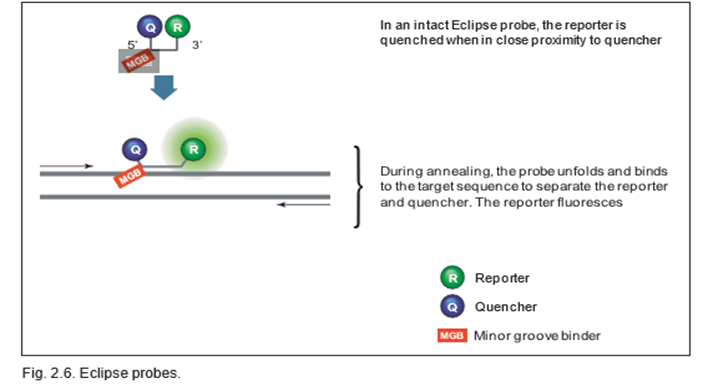
Eclipse probe .b:
نشانگر qPCR با استفاده از پروب Eclipse از دو آغازگر و یک پروب الیگونوکلئوتیدی خاص توالی استفاده می کند. این نشانگر، مکمل یک دنباله در آمپلیکون است و حاوی یک گزارشگر فلورسنت در انتهای 3’، یک خاموش کننده در انتهای 5′ و یک شیار چسبنده کوچک است. کاوشگر هیبرید نشده ترکیبی را اتخاذ می کند که گزارشگر و خاموش کننده را به هم نزدیک کند و گزارشگر را خاموش می شود. در طول مرحله (annealing) PCR، پروب با کمک بایندر شیار کوچک به هدف هیبرید می شود. بنابراین شکل کاوشگر خطی می شود و گزارشگر و خاموش کننده از هم جدا شده و به گزارشگر اجازه می دهد فلورسانس آزاد کند.
Amplifluor chemistry .c:
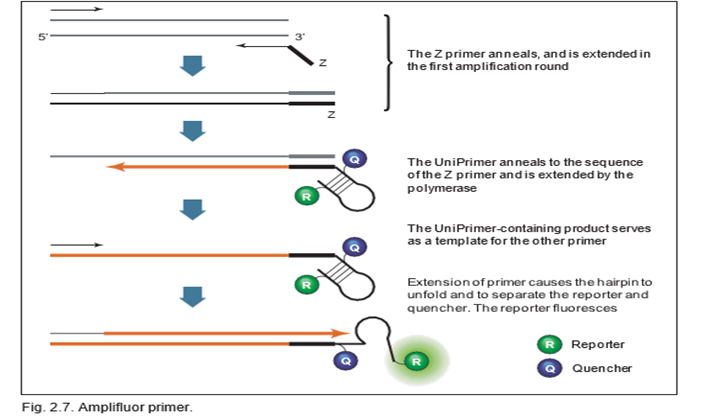
در سنجش qPCR با استفاده از نشانگر آمپلیفلور از دو پرایمر اختصاصی هدف و یک پرایمر یونیورسال به نام UniPrimer استفاده می شود. اولین پرایمر خاص الگوی هدف، شامل یک دنباله گسترش ‘5 بنام دنباله Z است که در انتهای ‘3 از UniPrimer نیز یافت می شود. همانطور که در شکل نشان داده شده است، UniPrimer یک ساختار سنجاق سر را تشکیل می دهد و یک گزارشگر فلورسنت و یک خاموش کننده به ترتیب به انتهای ‘5 و ‘3 ساختار ساقه متصل می شوند. در ترکیب سنجاق سر، فلورسانس گزارشگر به دلیل نزدیکی به کوئنچر خاموش می باشد. در طول اولین چرخه تکثیر، اولین آغازگر مخصوص الگوی هدف (با دنباله Z) با الگوی هدف هیبرید می شود و تکثیر می یابد. در طول دومین چرخه تکثیر، دومین پرایمر اختصاصی هدف برای سنتز یک الگوی هدف جدید، که حاوی دنباله مکمل Z است، تکثیر می شود. محصول چرخه تکثیر دوم می تواند به عنوان الگوی UniPrimer عمل کند. در سومین چرخه تکثیر، UniPrimer توسعه یافته به عنوان یک الگو برای چرخه تکثیر بعدی عمل می کند. در چرخه چهارم، گسترش الگو از طریق ناحیه سنجاق سر UniPrimer باعث می شود که UniPrimer باز شود و یک پیکربندی خطی اتخاذ کند که به گزارشگر اجازه می دهد فلورسانس آزاد کند. امپلیفای شدن نمایی، با استفاده از پرایمر اختصاصی دوم برای الگوی هدف و UniPrimer در چرخه های تکثیر بعدی رخ می دهد.
Scorpions’ primer .d:
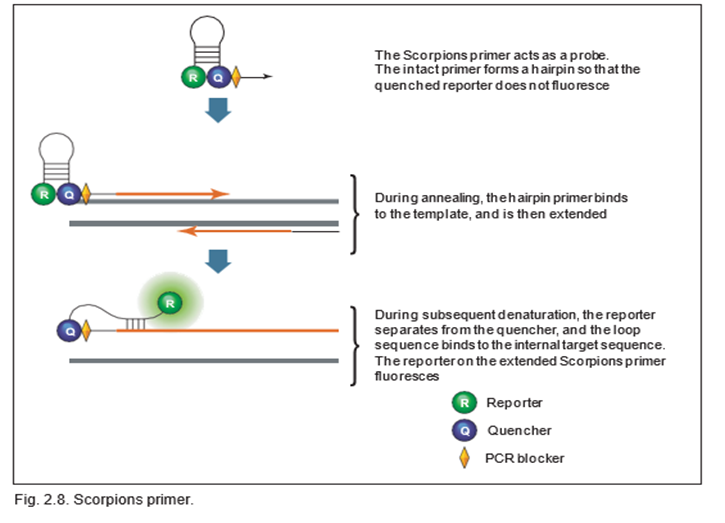
در واکنشهای حاوی پرایمرهای Scorpions از دو پرایمر استفاده میشود، که یکی از آنها مانند پروب عمل میکند و شامل یک ساختار حلقه-ساقه ای با یک گزارشگر فلورسنت در سمت ‘5 و یک خاموش کننده (کوانچر) در سمت ‘3 می باشد (همانطور که در شکل زیر نشان داده شده است). دنباله حلقه پروب درScorpions، مکمل بخش داخلی دنباله ی الگوی هدف در همان رشته است. در طول اولین چرخه تکثیر، پرایمر Scorpions گسترش می یابد و توالی مکمل دنباله حلقه روی همان رشته گسترده می شود. پروب Scorpions حاوی یک مسدود کننده PCR در انتهای ‘3 کوئنچر می باشد و مانع از رونویسی رشته ی مقابل الگوی هدف می شود. پس از دناتوره شدن و بازپخت بعدی، حلقه پروب Scorpions با یک برهمکنش درون مولکولی به دنباله هدف هیبرید می شود و گزارشگر از خاموش کننده جدا می شود.
LUX primer assays .e:
واکنش حاوی پرایمر LUX از دو پرایمر استفاده میکند که یکی از آنها یک پرایمر به شکل سنجاق مو و دیگری یک گزارشگر فلورسنت متصل به انتهای ‘3 است. در پرایمر دست نخورده (خاموش)، گزارشگر توسط ساختار ثانویه سنجاق سری خاموش می شود. در طول امپلیفیکیشن، پرایمر LUX با محصول نهایی ترکیب می شود و پروب گزارشگر فلورسانس آزاد می کند.
BD QZyme primers .f:
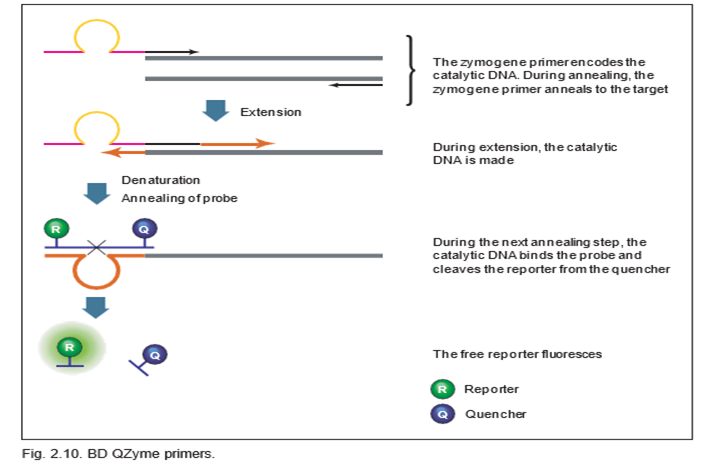
در واکنش qPCR حاوی BD QZyme primer، از یک پرایمر زیموژن اختصاصی هدف، یک پرایمر رشته ی ریورس (معکوس) و یک بستر الیگونوکلئوتیدی یونیورسال استفاده میکنند. الیگونوکلئوتید حاوی یک پروب فلورسنت در انتهای ‘5 و یک خاموش کننده (کوئنچر) در انتهای ‘3 است. هنگامی که بستر الیگونوکلئوتیدی دست نخورده است، فلورسانس گزارشگر به دلیل نزدیکی با کوئنچر، خاموش می شود. پرایمر zymogene حاوی دنباله ای است که یک DNA کاتالیزوری را کد می کند. در طول اولین چرخه تکثیر، پرایمر zymogene گسترش می یابد. در چرخه دوم، محصول چرخه اول به عنوان الگو توسط پرایمر معکوس استفاده می شود، که برای ایجاد یک توالی هدف جدید حاوی یک منطقه DNA کاتالیزوری تکثیر می یابد. در مرحله بازپخت بعدی، سوبسترای الیگونوکلئوتیدی نشاندار شده با فلورسنت به دنباله DNA کاتالیزوری هیبرید شده و جدا می شود. در نتیجه این جدا شدن موجب میشود پرایمر از خاموش کننده جدا شده و در نتیجه یک سیگنال فلورسنت متناسب با مقدار محصول تقویت شده در نمونه ایجاد شود.
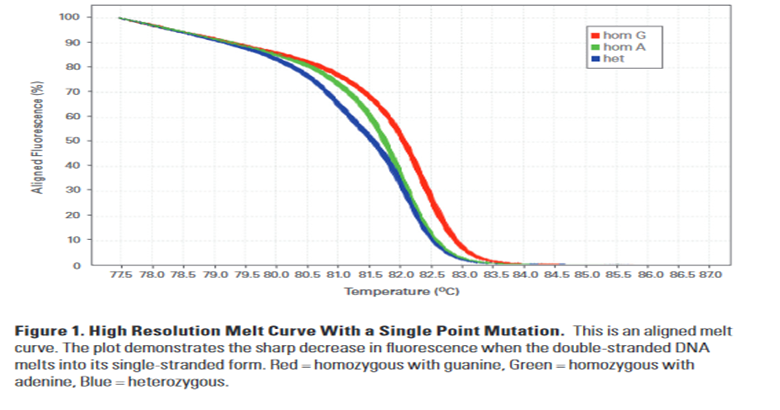
HRM:
تجزیه و تحلیل ذوب با وضوح بالا (HRM) یک روش جدید تجزیه و تحلیل پس از PCR است که برای شناسایی تنوع ژنتیکی در توالی های اسید نوکلئیک استفاده می شود و بسیار روش ساده و سریعی است. این روش مبتنی بر تکنیکهای منحنی ذوب (تفکیک) PCR است و با در دسترس بودن اطلاعات رنگهای پیوند دهنده DNA دو رشتهای (dsDNA) و بهره گیری از نرمافزارها و دستگاه های دقیق و آنالیزداده های PCR در زمان واقعی در هر تست اطلاعات کاملی را بدست می دهد. در واقع تجزیه و تحلیل HRM میتواند توالیهای DNA را بر اساس ترکیب، طول، محتوای GC یا مکمل رشتهای متمایز کند. تجزیه و تحلیل HRM با تجزیه و تحلیل منحنی مذاب استاندارد از سه جهت متفاوت است:
- شیمیایی: تجزیه و تحلیل HRM از رنگ های روشن تر در غلظت های بالاتر استفاده می کند.
- ابزارها: تجزیه و تحلیل HRM به ابزارهایی نیاز دارد که جمع آوری کننده ی داده های فلورسانس در وضوح دمایی بهتر می باشد.
- نرم افزار: تجزیه و تحلیل HRM به نرم افزار پیچیده تری نیاز دارد که از الگوریتم های مقیاس بندی فلورسنت جدید استفاده می کند و تجزیه و تحلیل HRM را با پیشرفت PCR در ناحیه مورد نظر در حضور رنگ اتصالی dsDNA آغاز می کند. این رنگ قابل اتصال، فلورسانس بالایی در هنگام اتصال به dsDNA و فلورسانس کم در حالت غیر متصل دارد. پیشرفت واکنش با یک مرحله ذوب با وضوح بالا، با استفاده از ابزار دقیقی که قادر به ثبت تعداد زیادی از نقاط داده فلورسنت در هر تغییر دما با دقت بالا می باشد، دنبال و ثبت می شود. هنگامی که dsDNA به رشته های منفرد تجزیه می شود (یا ذوب می شود)، رنگ آزاد می شود و باعث تغییر در فلورسانس می شود. بنابراین نتیجه ی نمایه منحنی مذاب مشخصه آمپلیکون است.
تجزیه و تحلیل HRM نیازی به پردازش دستی پس از PCR ندارد، در یک سیستم لوله بسته انجام می شود و هزینه واکنش پایینی نسبت به سایر موارد دارد.
تجزیه و تحلیل HRM:
در تجزیه و تحلیل HRM، ناحیه مورد نظر با PCR در حضور رنگ اتصال فلورسنت dsDNA تقویت می شود. پس از PCR، محصول به تدریج ذوب می شود و فلورسانس منتشر شده با یک ابزار تخصصی اندازه گیری می شود تا یک منحنی مشخص ایجاد شود. منحنی مذاب ایجاد شده ویژگی هایی مانند محتوایGC، طول قطعه، توالی و ماهیت هتروزیگوسیتی بودن نمونه را برای هر آمپلیکون نمایش می دهد. پروفایل های به دست آمده می توانند اطلاعات ارزشمندی را برای غربالگری جهش، ژنوتیپ، متیلاسیون و سایر کاربردهای تحقیقاتی ارائه دهند. تجزیه و تحلیل HRM از دو مشاهدات نمایه استفاده می کند:
1. منحنی های مذاب که از نظر شکل مشابه هستند اما با تفاوت دمای ذوب (Tm) آمپلیکون از یکدیگر قابل تشخیص هستند. بطور معمول چنین پروفایل هایی توسط نمونه های هموزیگوت که با نمونه های نوع وحشی مقایسه می شوند، تولید می شوند. در چنین شرایطی، اختلاف Tm بین نمونهها به دلیل تغییر توالی در نمونه های نوع وحشی دیده می شود.
2. منحنی های مذاب که یک شکل منحنی متمایز از منحنی های مذاب هموزیگوت را نشان می دهند. این پروفایل ها معمولاً به دلیل وجود عدم تطابق جفت پایه (هترودپلکس) موجود در مخلوط محصول PCR است.
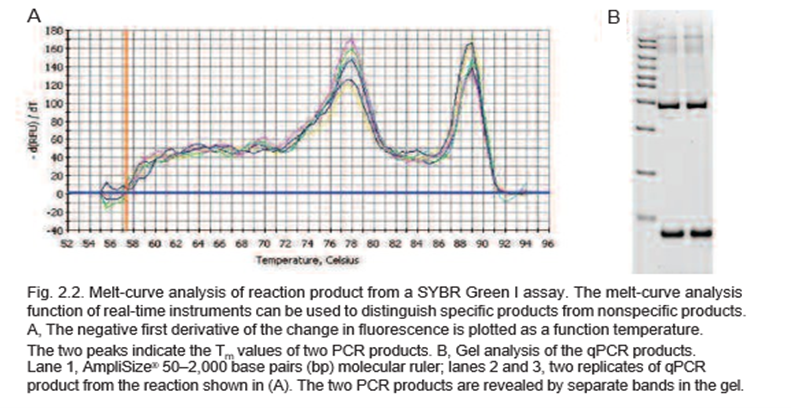
تجزیه و تحلیل منحنی مذاب می تواند برای شناسایی محصولات مختلف واکنش، از جمله محصولات غیر اختصاصی استفاده شود. بنابراین پس از اتمام واکنش، منحنی مذاب با افزایش دما در مراحل کوچک و نظارت بر سیگنال فلورسنت در هر مرحله ثبت می شود. با دناتوره شدن dsDNA در واکنش (یعنی با ذوب شدنDNA)، فلورسانس کاهش می یابد و اولین مشتق منفی حاصل از تغییر در فلورسانس به عنوان تابعی از دما رسم می شود. یک پیک مشخص در دمای ذوب آمپلیکون (دمایی که در آن 50 درصد از جفتهای باز یک DNA دوبلکس جدا میشوند) آنرا از سایر محصولات مانند پرایمر-دایمرها که در دماهای مختلف ذوب میشوند متمایز میکند. برای درک بهتر به تفسیر شکل زیر دقت نمایید. در تصویر زیر، پیک مذاب با Tm 89 درجه سانتیگراد محصول خاص را نشان می دهد و مربوط به نوار بالایی در خطوط 2 و 3 روی ژل است. پیک با Tm 78 درجه سانتی گراد محصول غیراختصاصی را نشان می دهد و مربوط به نوار پایینی در خطوط 2 و 3 روی ژل است.
اشکال عمده رنگ های متصل شونده بهDNA، عدم اختصاصیت آنها است، یعنی رنگ های متصل شونده به DNA به همه ی رشته های dsDNA متصل می شوند. در نتیجه حضور محصولات غیر اختصاصی در یک واکنش PCR در زمان واقعی ممکن است در میزان جذب فلورسانس کلی تاثیر گذاشته و دقت کمّیسازی را کاهش دهد. پیامد دیگر این است که رنگ های متصل شونده به DNA را نمی توان برای واکنش های چندگانه استفاده کرد زیرا سیگنال های فلورسنت از آمپلیکون های مختلف قابل تشخیص نیستند.
در عوض، می توانید با استفاده از SYBR Green I و واکنشهای موازی، بیان ژنهای متعدد (مانند ژن مورد نظر و ژن مرجع) را در یک آزمایش PCRدر زمان واقعی بررسی کنید.
مزایای Real-time PCR:
- قابلیت نظارت بر پیشرفت واکنش PCR در زمان واقعی تکثیر
- قابلیت اندازه گیری دقیق مقدار آمپلیکون در هر سیکل، که امکان کمّی سازی بسیار دقیق مقدار ماده اولیه در نمونه ها را فراهم می کند
- افزایش دامنه دینامیکی تشخیص
- تکثیر و تشخیص در یک لوله انجام می شود و دستکاری پس از PCR را حذف می کند.
References:
- I Made Artika, Y. Permata Dewi, I.M. Nainggolan, J.E. Siregar & U. Antonjaya. “Real-Time Polymerase Chain Reaction: Current Techniques, Applications, and Role in COVID-19 Diagnosis”. Genes (Basel). 2022 Dec; 13(12): 2387. Published online 2022 Dec 16. Doi: 10.3390/genes13122387.
- Real-time PCR handbook. Life Technologies. www.gene-quantification.de
- A Guide to High Resolution Melting (HRM) Analysis. www.appliedbiosystems.com
- Real-Time PCR Applications Guide, Bio-Rad:
- Mathews DH et al., Expanded sequence dependence of thermodynamic parameters provides robust prediction of RNA secondary structure, J Mol Biol 288, 910–940 (1999))
- Rozen S and Skaletsky HJ, Primer3 on the WWW for general users and for biologist programmers, pp 365–386 in Bioinformatics Methods and Protocols: Methods in Molecular Biology (Krawetz S and Misener S, eds), Humana Press, Totowa, NJ (2000); source code available at http://fokker.wi.mit.edu/primer3/
- Zuker M et al., Algorithms and thermodynamics for RNA secondary structure prediction: a practical guide, pp 11–43 in RNA Biochemistry and Biotechnology (Barciszewski J and Clark BFC, eds), Kluwer Academic Publishers, Boston (1999)
- Real-Time PCR Applications Guide. Degen, Hans-Joachim, Deufel, Annette, Eisel, Doris Grünewald-Janho, Stefanie Keesey, Joe. 2006 by Roche Diagnostics GmbH, Mannheim. PCR Applications Manual 3rd edition. www.Roche-Applied-Science.com
- Quantitative Real-Time PCR Methods and Protocols (3rd Edition). Editors: Roberto Biassoni Laboratorio Medicina Molecolare Dipartimento Medicina Traslazionale Istituto Giannina Gaslini Genova, Italy. ISSN 1064-3745 ISSN 1940-6029 (electronic) ISBN 978-1-4939-0732-8 ISBN 978-1-4939-0733-5 (eBook) DOI 10.1007/978-1-4939-0733-5 Springer New York Heidelberg Dordrecht London Library of Congress Control Number: 2014936291 © Springer Science+Business Media New York 2014.





